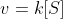Michaelis-Menten-Kinetik von Alpha-Amylase
α-Amylasen spalten Amylose (neben Amylopektin einem Bestandteil von Stärke), wodurch unter anderem Maltose entsteht. Durch Nachweis der Stärke mittels Lugol’scher Lösung und Quantifizierung über UV/VIS Spektroskopie lässt sich die Enzymkinetik bestimmen, wodurch die Michaelis-Menten-Parameter bestimmt werden können.
Allgemeine Hinweise zum Experimentieren und Disclaimer beachten!
Einleitung
α-Amylasen spalten Amylose (neben Amylopektin einem Bestandteil von Stärke), wodurch unter anderem Maltose entsteht. Durch Nachweis der Stärke mittels Lugol’scher Lösung und Quantifizierung über UV/VIS Spektroskopie lässt sich die Enzymkinetik bestimmen, wodurch die Michaelis-Menten-Parameter bestimmt werden können.
Schwierigkeitsgrad
Schülerexperiment - schwer
Geräte
50 mL Becherglas (2 x), 100 mL Messkolben, Reagenzglas-Ständer, 15 mL Falcon Tubes, Mikroliterpipetten (5000 µL, 1000 µL & 10 µL), Stoppuhr, Magnetrührer mit Heizplatte und Rührfisch, Küvetten (Halbmikro), UV/VIS-Spektrometer, Mikrowelle, Analysenwaage, Computer mit MS Excel (oder anderweitiges Tabellenkalkulationsprogramm)
Chemikalien
lösliche Stärke
α-Amylase aus Bacillus amyloliquefaciens (> 250 U/g)
Lugol’sche Lösung
Durchführung
Zuerst wird eine 1 mg/mL Stärkelösung durch Einwiegen von 100 mg löslicher Stärke und Verdünnen auf 100 mL Wasser hergestellt. Die Stärke muss in der Siedehitze gelöst werden.
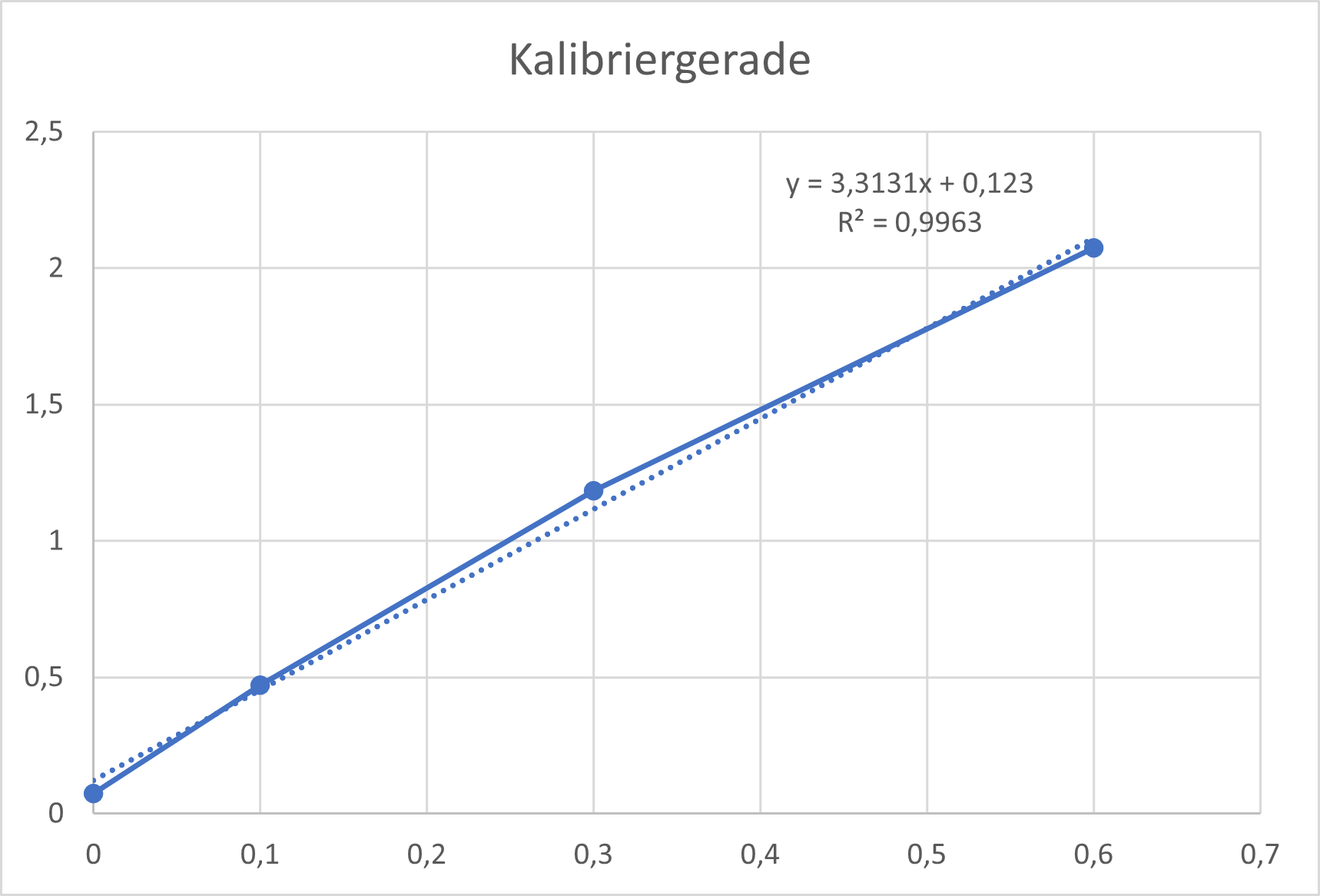
Zur Herstellung der Kalibriergeraden werden von dieser Stärkelösung 0, 100, 300 und 600 µL jeweils mit der entsprechenden Menge Wasser auf 1000 µL verdünnt und zur Gänze in eine Lösung aus 3 mL Wasser und einem Tropfen Iod-Kaliumiodidlösung überführt. Die Absorption bei 600 nm wird daraufhin zügig am UV/VIS-Spektrometer bestimmt.
Zur Reaktion werden 10 mL einer Stärkelösung mit einer Konzentration von 0,5, 0,75 und 1,0 mg/mL zusammen mit 1 µL einer verdünnten Amylase-Lösung angesetzt (10 µL Amylase auf 990 µL dest. Wasser). Nach 1, 3, 5, 10, 15, 30 und 45 min werden 1000 µL als Probe entnommen und wiederum in eine Lösung aus 1 Tropfen Iod-Kaliumiodid-Lösung in 3 mL dest. Wasser überführt. Die Absorption bei 600 nm wird daraufhin zügig am UV/VIS-Spektrometer bestimmt.
| Zeit [min] | Absorption | ||
|---|---|---|---|
| 1,00 mg/mL | 0,75 mg/mL | 0,50 mg/mL | |
| 1 | 2,182 | 1,461 | 1,095 |
| 3 | 1,816 | 1,194 | 0,910 |
| 5 | 1,467 | 0,901 | 0,716 |
| 10 | 1,087 | 0,618 | 0,501 |
| 15 | 0,826 | 0,467 | 0,422 |
| 30 | 0,591 | 0,278 | 0,320 |
| 45 | 0,472 | 0,217 | 0,280 |
Das kann natürlich auch grafisch dargestellt werden:
Zur Datenauswertung werden die Absorptions/Zeit-Verläufe mittels der Kalibriergerade in Konzentrations/Zeit-Verläufe umgerechnet:
| Zeit [min] | Konzentration [mg/mL] | ||
|---|---|---|---|
| 1,00 mg/mL | 0,75 mg/mL | 0,50 mg/mL | |
| 1 | 0,621 | 0,404 | 0,293 |
| 3 | 0,511 | 0,323 | 0,238 |
| 5 | 0,406 | 0,235 | 0,179 |
| 10 | 0,291 | 0,149 | 0,114 |
| 15 | 0,212 | 0,104 | 0,090 |
| 30 | 0,141 | 0,047 | 0,059 |
| 45 | 0,105 | 0,028 | 0,047 |
Danach erfolgt die Bestimmung der Anfangsgeschwindigkeit durch lineare Regression anhand der ersten drei Messpunkte:
Entsorgung
Alle Lösungen, die Iod bzw. Kaliumiodid enthalten, werden als halogenhaltige Salzlösung entsorgt.
Erklärung
Was versteht man unter der “Geschwindigkeit” einer chemischen Reaktion?
Zur Beantwortung dieser Frage wird die folgende Reaktion betrachtet, bei der ein Substrat (S) irreversibel zu einem Produkt (P) umgewandelt wird:
Die Geschwindigkeit der Reaktion würde dann als Änderung der Produktkonzentration (oder negative Änderung der Substratkonzentration) pro Zeiteinheit definiert werden:
Die Reaktionsgeschwindigkeit steht über einen Proportionalitätsfaktor - der Geschwindigkeitskonstante - in Zusammenhang mit der Substratkonzentration:
Hierbei handelt es sich um eine Reaktion 1. Ordnung, da die Geschwindigkeitskonstante in einem linearen Zusammenhang mit der Substratkonzentration steht. Auch andere Reaktionsordnungen sind möglich, beispielsweise ist bei einer Reaktion 0. Ordnung die Geschwindigkeit unabhängig von der Konzentration der Ausgangssubstanz.
Die Reaktionsgeschwindigkeit muss experimentell ermittelt werden, indem im Verlauf der Reaktion die Zunahme des Produkts (oder Abnahme des Edukts) beispielsweise über die Änderung der Absorption gemessen wird. Dabei zeigt sich, dass sich die Konzentration anfangs schnell ändert, sich aber irgendwann einer Gleichgewichtskonzentration annähert - die Reaktionsgeschwindigkeit ist also über den Reaktionsverlauf nicht konstant.
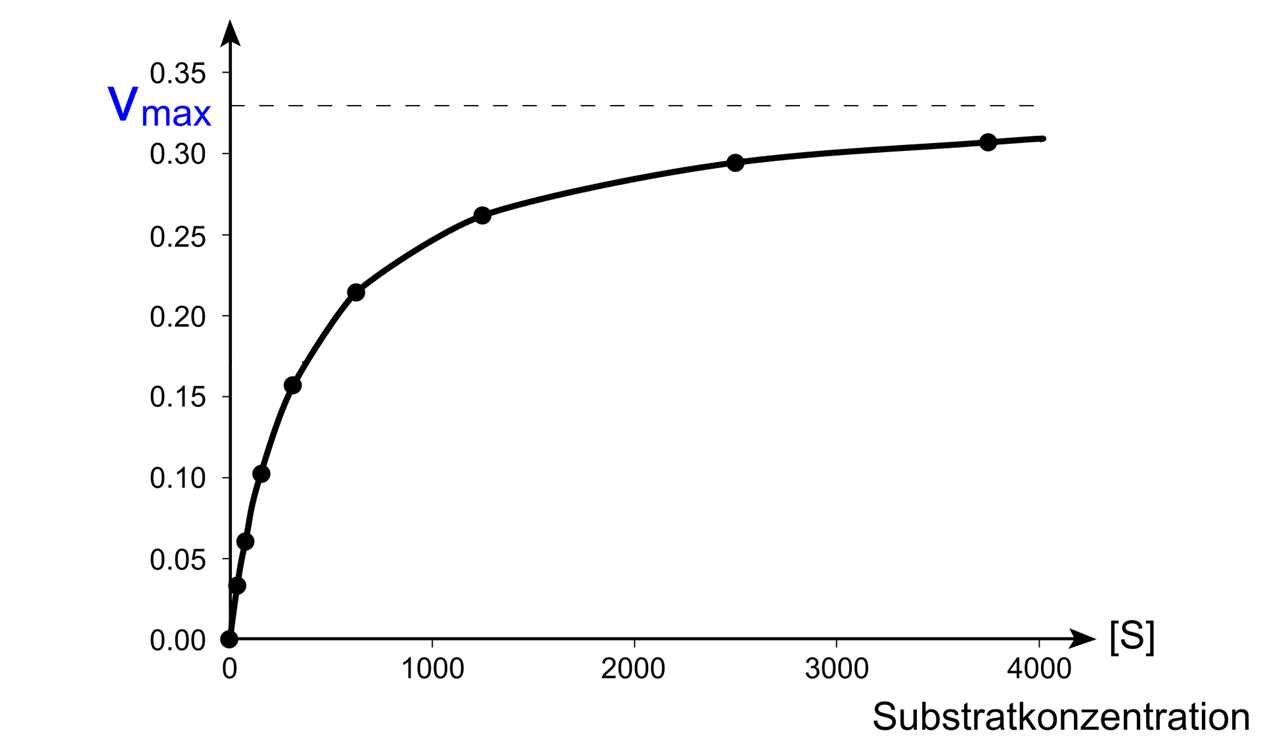
Wird nun die Anfangsgeschwindigkeit von enzymatisch katalysierten Reaktionen bei unterschiedlichen Substratkonzentrationen gemessen, zeigt sich, dass die Anfangsgeschwindigkeit bei steigender Substratkonzentrationen anfangs linear zunimmt, bis sie sich irgendwann einer Maximalgeschwindigkeit vmax annähert:
Zur Beschreibung dieser kinetischen Eigenschaft schlugen Leonor Michealis und Maud Menten 1913 ein einfaches Modell vor. Hierbei formulierten sie einen Enzym-Substrat-Komplex, der als Zwischenprodukt auftreten muss:
Durch drei Annahmen kann das Modell vereinfacht werden:
Die Reaktion wird zum Zeitpunkt t ≈ 0 betrachtet, wodurch noch kein Produkt gebildet wurde und der Enzym-Substrat-Komplex irreversibel zum Produkt zerfällt
Die Konzentration des Enzym-Substrat-Komplexes ist konstant, dessen Bildung und Zerfall also gleich schnell (Steady-State)
Die Konzentration des Substrats ist deutlich höher als jene des Enzyms, die Konzentration an im Enzym gebundenem Substrat ist folglich gegenüber der Gesamtsubstratkonzentration vernachlässigbar.
Dadurch vereinfacht sich die Reaktionsgleichung zu folgendem Ausdruck und die Michealis-Menten-Gleichung lässt sich daraus ableiten (zur exakten Herleitung sei auf einschlägige Literatur verwiesen):
Die Michaelis-Menten-Konstante gibt folglich die Substratkonzentration an, bei der die Reaktionsgeschwindigkeit die Hälfte ihres Maximalwertes erreicht. KM ist ein wichtiges Charakteristikum enzymatisch katalysierter Reaktionen und insbesondere für die biologische Funktion der Enzyme von Bedeutung.
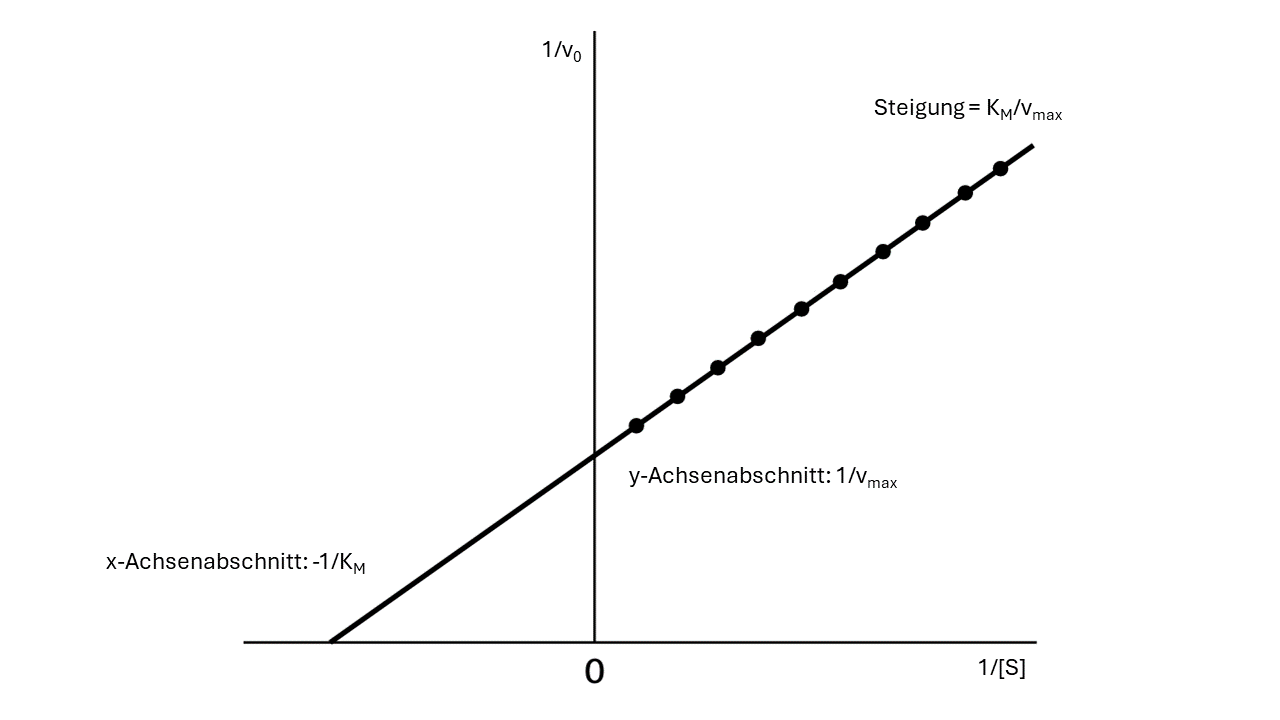
Doch wie werden KM und vmax nun bestimmt? Dazu wird die Michaelis-Menten-Gleichung zuerst auf ihre doppelt-reziproke Darstellung umgeformt, wodurch eine Geradengleichung erhalten wird:
Video
Literatur
Cochran et al. "Kinetic Analysis of Amylase Using Quantitative Benedict's and Iodine Starch Reagents" J. Chem. Educ. 85, 3 (2008): 401
Berg, Jeremy M., and John L. Tymoczko. Stryer biochemie. Vol. 8. Heidelberg: Springer Spektrum, 2018.
Gefährdungsbeurteilung (Gestis-Stoffdatenbank)
Lösliche Stärke (kein Gefahrstoff nach GHS)
Werbung
Diese Anzeige wird von unseren Werbepartnern geschaltet und hilft uns dabei, die Nutzung unserer Website für Sie weiterhin kostenlos zu halten. Durch das Anzeigen dieser Werbung unterstützen Sie direkt unsere Inhalte und ermöglichen es uns, die Kosten für den Betrieb unserer Website zu decken.
DNA-Extraktion aus Bananen
Nicht nur in Filmen und TV-Serien werden Täter oft anhand einer DNA-Spur überführt. Doch wie viel DNA steckt eigentlich in jedem Kern unserer Zellen und wie extrahiert man diese am einfachsten? Ein einfacher Versuch am Beispiel einer Banane demonstriert dies eindrucksvoll.
Allgemeine Hinweise zum Experimentieren und Disclaimer beachten!
Einleitung
Nicht nur in Filmen und TV-Serien werden Täter oft anhand einer DNA-Spur überführt. Doch wie viel DNA steckt eigentlich in jedem Kern unserer Zellen und wie extrahiert man diese am einfachsten? Ein einfacher Versuch am Beispiel einer Banane demonstriert dies eindrucksvoll.
Schwierigkeitsgrad
Schülerexperiment - einfach
Geräte
Becherglas 100mL, Erlenmeyerkolben 100mL, Mörser und Pistill, Glasspatel, Trichter, Filterpapier
Chemikalien
Ethanol
Durchführung
Eine Lösung aus 5 mL Spülmittel, 45mL Wasser sowie einem halben Teelöffel Salz wird in einem 100 mL Becherglas bereitet. Zu dieser wird ca. ein ¼ einer Banane gegeben, welches zuvor in einem Mörser mittels Pistill zerdrückt wurde. Die zähflüssige Masse wird mit einem Glasspatel kräftig umgerührt und anschließend dieses Gemisch in den Erlenmeyerkolben filtriert.
Nun werden ca. 25 mL gekühlter Ethanol hinzugefügt. Schon nach kurzer Zeit bilden sich DNA-Polymere, die zur Oberfläche der Flüssigkeit aufsteigen.
Entsorgung
Alle Feststoffe und anfallende Lösungen können im Restmüll bzw. dem Ausguss entsorgt werden.
Erklärung
Beim Zerdrücken der Banane im Mörser werden die Zellen auseinandergerissen, die Zellwände teilweise zerstört und das Innere der Zelle freigesetzt. Durch das Hinzufügen des Spülmittels werden auch die restlichen Zellwände und die Zellmembran aufgelöst und die DNA freigelegt. Dies geschieht da Spülmittel ein Detergenz – ein Stoff der Fette in Lösung bringt – ist und somit die aus Fetten bestehenden Zellwände und Zellmembranen lösen kann.
Durch die Polarität von Wasser löst dieses die negativ geladene DNA, wobei der Effekt durch die Tatsache, dass die Salzkonzentration der Lösung ungefähr der im lebenden Organismus entspricht verstärkt wird.
Der abschließend hinzugegebene Ethanol entfernt die Hydrathülle der DNA und verringert die Löslichkeit dieser - die DNA beginnt auszufallen und zu polymerisieren.
Fotos
Video
Diese Anzeige wird von unseren Werbepartnern geschaltet und hilft uns dabei, die Nutzung unserer Website für Sie weiterhin kostenlos zu halten. Durch das Anzeigen dieser Werbung unterstützen Sie direkt unsere Inhalte und ermöglichen es uns, die Kosten für den Betrieb unserer Website zu decken.
Gramfärbung
Die Gram Färbung – entwickelt vom dänischen Bakteriologen Hans Christian Gram ermöglicht es, Bakterien anhand von Unterschieden im Aufbau der Zellwände zu unterscheiden. Aufgrund der Einfachheit und kurzen Zeitdauer bis zu einem Ergebnis ist die Gram Färbung auch heute noch ein wichtiges Mittel in der Diagnostik bakterieller Infektionskrankheiten.
Allgemeine Hinweise zum Experimentieren und Disclaimer beachten!
Einleitung
Die Gramfärbung – entwickelt vom dänischen Bakteriologen Hans Christian Gram - ermöglicht es, Bakterien anhand von Unterschieden im Aufbau der Zellwände zu unterscheiden. Aufgrund der Einfachheit und kurzen Zeitdauer bis zu einem Ergebnis ist die Gramfärbung auch heute noch ein wichtiges Mittel in der Diagnostik bakterieller Infektionskrankheiten.
Schwierigkeitsgrad
Schülerexperiment - schwer
Geräte
Mikroskop, 250 ml Becherglas, 5x 50 ml Becherglas, Pasteurpipetten, Objektträger, Bunsenbrenner, Feuerzeug
Chemikalien
0,1 % ethanolische Kristallviolett Lösung
0,1 % wässrige Safranin-T Lösung
5 % Lugol’sche Lösung
Aceton
Immersionsöl
Hinweis
Kristallviolett und Safranin-T sind hervorragende Farbstoffe. Entstandene Flecken auf Kleidung oder Gegenständen lassen sich kaum mehr entfernen
Durchführung
Herstellung der benötigten Lösungen
0,1 % ethanolische Kristallviolett Lösung: 0,08 g Kristallviolett werden in 100,0 mL 96 % Ethanol gelöst.
0,1 % wässrige Safranin-T Lösung: 0,1 g Safranin T werden in 99,9 mL dest. Wasser gelöst.
5 % Lugol’sche Lösung: 5 g Iod und 10 g Kaliumiodid werden in einem 250 mL Becherglas eingewogen und mit 85 mL verdünnt.
Vorbereiten der Probe
Eine bakterienhaltige Probe (z.B. ein Tropfen Wasser aus einem Heuaufguss) wird auf einem Objektträger ausgestrichen, sodass die eingenommene Fläche ungefähr 1x1 cm entspricht und an der Luft zum Trocknen belassen. Zur Fixierung wird der Objektträger 3x durch die nicht leuchtende Brennerflamme gezogen.
Gramfärbung
Der Objektträger mit der fixierten Probe wird nun auf ein 250 mL Becherglas gelegt und die Gramfärbung entsprechend dieser Reihenfolge durchgeführt. Nach jeder Lösung ist gut mit dest. Wasser zu spülen!
0,1 % Kristallviolett Lösung (60 sek.)
5 % Lugol’sche Lösung (60 sek.).
Aceton (2 sek.)
0,1 % Safranin-T Lösung (30 sek.)
Der Objektträger wird an der Unterseite und den Rändern mit Küchenrolle abgewischt und zum endgültigen Trocknen für wenige Minuten an der Luft aufgelegt.
Interpretation
Das Präparat wird bei mind. 1000x Vergrößerung ohne Deckglas (!) unter Ölimmersion mikroskopiert. Gram-positive Bakterien erscheinen dunkelviolett, während gram-negative Arten hell bzw. zartrosa dargestellt werden.
Entsorgung
Die Färbelösungen und das Spülwasser werden im Becherglas, auf dem der Objektträger liegt, gesammelt und später als organisch-wässriger, halogenhaltiger Abfall entsorgt.
Erklärung
Gram-Positive Bakterien besitzen eine sehr dicke Zellwand die aus Peptidoglycanen (Murein) besteht. Diese Mureinhülle wird durch das Kristallviolett angefärbt und reagiert mit Lugol’scher Lösung zu einem Kompex. Aceton kann diesen Farbstoff aus der Mureinhülle nicht mehr herauswaschen.
Anders als gram-postive Bakterien, haben gram-negative nur eine sehr dünne Mureinhülle, die von einer Lipidschicht umgebeben ist. Aceton wirkt lipidlösend, wodurch das Kristallviolett ausgewaschen werden kann. Das Gegenfärben mit Safranin-T macht die ansonsten durchsichtigen Bakterien wieder sichtbar.
Das Gramverhalten ist nicht immer eindeutig. Es gibt Baktierien, die ihr Gramverhalten ändern (z.B. mit der Lebensdauer) oder auch gramunbestimmte Arten.
Fotos
Video
Literatur
Gefährdungsbeurteilung (Gestis-Stoffdatenbank, Carl-Roth, Merck)
wässrige 0,1 % Safranin-T Lösung (Kein Gefahrstoff nach GHS)
Werbung
Diese Anzeige wird von unseren Werbepartnern geschaltet und hilft uns dabei, die Nutzung unserer Website für Sie weiterhin kostenlos zu halten. Durch das Anzeigen dieser Werbung unterstützen Sie direkt unsere Inhalte und ermöglichen es uns, die Kosten für den Betrieb unserer Website zu decken.